但是,我們如何嘗試加速我們識別某些目標效應的化合物的方式? 在這種情況下,這就是生成網絡領域迅速普及,以幫助使用諸如RNN之類的網絡生成新分子的地方。 目標是通過去除盡可能多的過程來加速發現階段的過程,我們必須通過重新藥物設計(de novo drug design)手動測試大量化合物來識別有效的化合物。
Project De Novo: Generating Novel Molecules
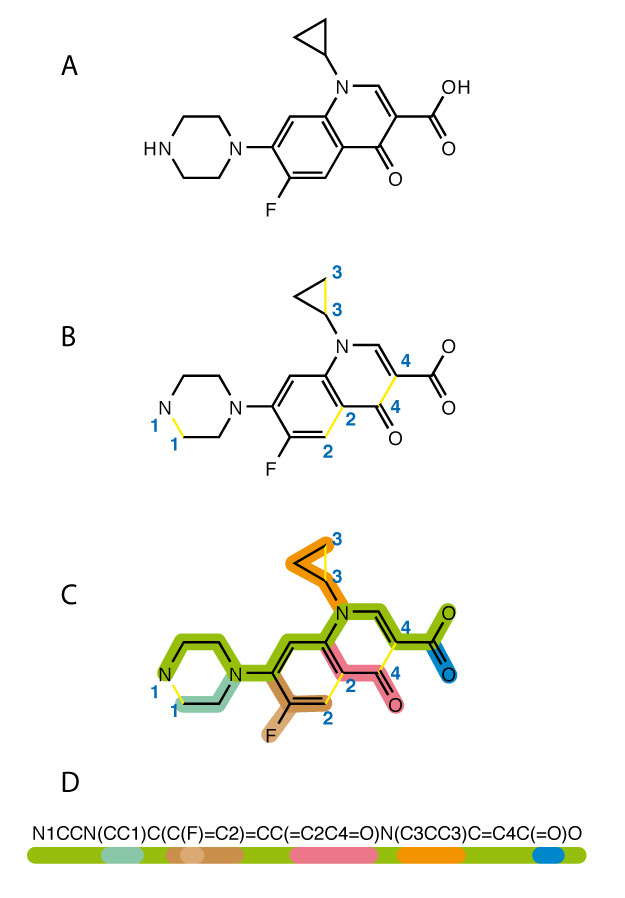
Each colour corresponds to a sequence of characters in a SMILES string
本文目標是使用遞歸神經網絡(RNN)生成新的分子。 De novo僅僅意味著從更簡單的分子合成新的複雜分子。 然而,有一個主要的警告:我們不知道這些分子有多麼有用,甚至它們的正確性,但在本文的最後會提到我們可以擴展這個模型的方法來做到這一點。 我們的想法是訓練模型學習SMILES字串中的模式,以便生成的輸出可以匹配有效的分子。 SMILES只是根據結構和不同組成的分子表示成字串(String),並且有助於讓電腦理解分子表示式。
from rdkit import Chem
from rdkit.Chem import Draw
import matplotlib.pyplot as plt
%matplotlib inline
penicillin_g_smiles = 'CC1([C@@H](N2[C@H](S1)[C@@H](C2=O)NC(=O)Cc3ccccc3)C(=O)O)C'
penicillin_g = Chem.MolFromSmiles(penicillin_g_smiles)
Draw.MolToMPL(penicillin_g, size=(200, 200))參考
https://chemistry.stackexchange.com/questions/43299/is-there-a-way-to-use-free-software-to-convert-smiles-strings-to-structures/57358
此外,有個網站也可以將化學結構翻譯成SMILES字串:
https://cactus.nci.nih.gov/
由於我們將文本作為數據提供到RNN神經網絡,因此RNN是用於生成新SMILES字符串的最佳網絡。 大多數論文和研究人員建議使用LSTM增強RNN的內部結構,LSTM是一種更有效訓練文本的神經網路。 因此,我們將使用2 LSTM層,在100,000個SMILES字符串的數據集上進行訓練。
Stage 1 — Mapping
該項目的第一階段是創建一個將字元映射到整數,以便RNN可以處理數據,反之,也可以將結果和輸出轉換回字元時。 我們需要創建一組所有獨特字串,並enumerate(為每個字元定義一個數值)每個項目。 SMILES字串由兩種類型的字元組成,它們是特殊字元,如“/”或“=”,以及元素符號,如“P”,“Si”,“Mg”等。我們將這些enumerate的獨特字元放入一個 獨特的字典,
unique_chars = sorted(list(set(raw_text)))
char_to_int = dict((c, i) for i, c in enumerate(unique_chars))
char_to_int.update({-1 : "\n"})
這裡合併“\ n”表示為.txt文件中的新行。
Stage 2— Data Preprocessing
一旦我們為字元映射創建了字典,我們就會呼叫字典char_to_int將SMILES字串數據集中的每個字元轉換為整數。 Normalization是將字元的每個整數值除以數據集中獨特字元的數量。 然後,我們使用NumPy將輸入陣列X reshape為[樣本,時間步長,生理化學特徵]的三維陣列,這是循環模型的預期輸入格式。 輸出變量Y是one-hot encoding,以在訓練模型後生成新的SMILES。 One-hot encoding適用於整數表示,並且刪除整數編碼變量,並為每個獨特的整數值添加一個新的二進制(0或1)變量。
Stage 3— Model Architecture

The Model Architecture, consisting of two LSTM layers with 256 units (Gupta et al.)
這個系統結構的設計是根據Gupta等人的研究論文“De Novo Drug Design的Generative Recurrent Networks”。採用這種架構,因為它在創建有效的SMILES字串時產生了97%的準確率,而且它是一個非常簡單的架構。它由兩個LSTM層組成,每個層都有一個隱藏的狀態向量H,它透過一個單元傳遞先前的資訊給下一個單元,這是RNN以前從未見過的。像這樣的更多循環連接允許網絡理解SMILES序列的更複雜的依賴性。這些層上使用0.25的壓差正則化,接著是使用softmax激活函數的neutron組成密集輸出層。 Softmax是一個函數,它將K個實數的向量(在我們的例子中是輸出向量Y)作為輸入,並將其normalize為由K個概率組成的概率分佈。
# Create the model (simple 2 layer LSTM)
model = Sequential()
model.add(LSTM(128, input_shape=(X.shape[1], X.shape[2]), return_sequences = True))
model.add(Dropout(0.25))
model.add(LSTM(256, return_sequences = True))
model.add(Dropout(0.25))
model.add(LSTM(512, return_sequences = True))
model.add(Dropout(0.25))
model.add(LSTM(256, return_sequences = True))
model.add(Dropout(0.25))
model.add(LSTM(128))
model.add(Dropout(0.25))
model.add(Dense(Y.shape[1], activation='softmax'))
隨意添加更多層或更改dropout rate,但請記住,在NN(神經網絡)中有更多層和神經元,在計算上它獲得的強度和準確度越高。
Stage 4— Training:
這裡的神經網絡使用categorical cross entropy作為loss function,以及Adam Optimizer。 包含100,000個SMILES字串的數據集=訓練了10個epochs的模型以及512 batch_size來學習。 這裡的經驗法則是更多的epochs +更小的batch_size =更好地理解數據,但代價是需要更長的訓練時間。 我們還可以利用檢查點(checkpoints),它是Keras函式庫中的內建功能來保存我們的訓練進度,以及每個epoch的模型權重,以便稍後傳輸或保存。
# Define checkpoints (used to save the weights at each epoch, so that the model doesn't need to be retrained) filepath="weights-improvement-{epoch:02d}-{loss:.4f}.hdf5"checkpoint = ModelCheckpoint(filepath, monitor = 'loss', verbose = 1, save_best_only = True, mode = 'min') callbacks_list = [checkpoint] # Fit the model model.fit(X, Y, epochs = 19, batch_size = 512, callbacks = callbacks_list) """TO TRAIN FROM SAVED CHECKPOINT""" # Load weights model.load_weights("weights-improvement-75-1.8144.hdf5") # load the model new_model = load_model ("model.h5") assert_allclose(model.predict(x_train),new_model.predict(x_train), 1e-5) # fit the model checkpoint = ModelCheckpoint(filepath, monitor='loss', verbose=1, save_best_only=True, mode='min') callbacks_list = [checkpoint]new_model.fit(x_train, y_train, epochs = 100, batch_size = 64, callbacks = callbacks_list)
Stage 5 — Generating New Molecules:
生成新分子相當簡單。 首先,我們加載預先訓練的權重,這樣我們就可以避免每次在生成新的SMILES字串之前訓練模型。 然後,我們從數據集中隨機選擇一個SMILES字串作為參考字串,並在範圍內生成指定數量的字元,並將整數值轉換回字元。 上面的代碼在技術上可以用於生成任何類型的文本,無論是故事,音樂還是SMILES分子。 根據自定義調整,學習速率,數據集大小和quality,結果會有所不同,但最後我們應該得到一種有效的分子表示字串。 在進行學習新的子結構和分支(N1CCN(CC1)C(C(F)=C2…)分子之前,模型可以藉由僅生成一個字符(NNNNNN)的序列開始。
這是分子二維結構 (O1C=C[C@H([C@H]1O2)c3c2cc(OC)c4c3OC(=O)C5=C4CCC(=O)5)

和三維結構:
O1C=C[C@H]([C@H]1O2)c3c2cc(OC)c4c3OC(=O)C5=C4CCC(=O)5
Possible Improvements
1. Checking the Validity of these Molecules:
我們不知道我們生成的分子是否具有任何用例, 我們必須藉由用於訓練的原始分子進行比較來驗證這些生成的分子是否是有效的分子結構。 藉由計算數據的共同生理化學特徵,以及對訓練數據的特徵使用主成分分析(Principal Component Analysis),我們可以確定新生成的分子是否相應地進行了轉換。
2. Are SMILES Strings even the most optimal way to represent molecules?:
如何確信LSTMs可以在最好的情況下生成文本的樣本,因為SMILES字串只是查看分子化學組成的基本方法。 我們可能會創建更好的方法來表示分子的複雜性,以便將來進行更準確的測試。 然而,目前,SMILES Strings和RNN是使用機器學習產生分子的規範。
3. The Dataset: Through data augmentation
我們可以採用不同SMILES字串的排列,並將它們添加到我們的數據集中。 增加數據集的另一種方法是enumerate SMILES字符串超出其原始形式,或以其他方式編寫。

Changing Drug Discovery: Fragment-Based Drug Discovery (FBDD)

這種產生分子的方法的主要用途之一是基於片段的藥物發現或FBDD。 這是在不使用哨兵(使用其存在作為終止條件的特殊值或特徵)開始輸入的地方,我們可以從已知結合目標疾病機制的片段開始。 通過將這個片段的SMILES字串作為輸入,我們可以用RNN逐漸“生長”分子的其餘部分,從而產生更有效地抵消某種功能的分子! 這種方法肯定有很多希望,並且看起來將來會被更多地使用會很有趣。
Conclusion + Key Takeaways
RNN僅使用數據集上的少量參數來提供了一種簡單而有效的分子生成方法。 該領域的大多數研究都希望結合RL或對抗性訓練等結構,以幫助增加產生的有效分子的數量,或使模型偏向於創造具有特定藥物某些特性的分子。 希望在未來採用這種方法,基於片段的藥物發現變得更加普遍,並有助於使藥物開發更加可行和經濟。 AI是移除創造療法的猜測的關鍵。
Key Takeaways
1. 緩慢而昂貴的藥物開發和材料科學研究是目前方法進展緩慢的直接結果
2. 使用機器學習,我們可以提高研發的速度。
3. 機器學習用於生成分子的最有效用途是使用RNN和SMILES分子字符串表示,但它們不是最佳方式。
4. 我們還有很長的路要走,才能使用人工智能以更快的速度製作更準確有效的分子結構,但還有很多值得期待的東西!
https://towardsdatascience.com/generating-molecules-with-the-help-of-recurrent-neural-networks-c3fe23bd0de2

沒有留言:
張貼留言